分子シミュレーションとその応用
分子シミュレーションは、原子間に働く力をもとに原子の動きを計算する方法で、いろいろなところで応用されていますが、私たちは、溶媒の存在を考慮した分子動力学法(molecular dynamics, MD)を使って、タンパク質にリガンドが結合する過程を解析する研究、生体内で特定の構造に折りたたまれるフォールディングの過程を解析する研究、構造予測プログラムで作成した予測構造からシミュレーションをスタートさせ、構造を修正して、予測の精度をさらに高める研究、そのほか、タンパク質分子の機能を解析するためのさまざまな研究を行っています。本研究は、寺田透准教授に指導をお願いしています。
リガンド結合過程の解析
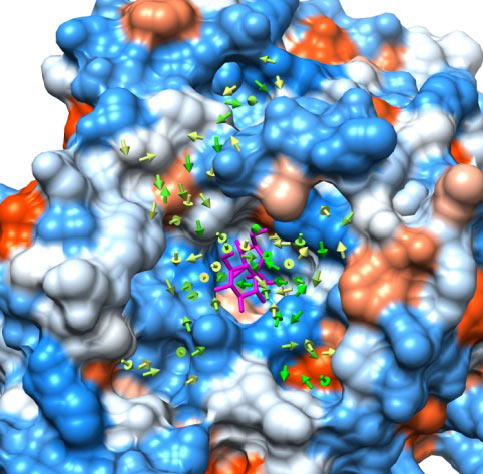
私たちは、粗視化モデルMARTINIを用いた分子動力学シミュレーションによるタンパク質-リガンド結合過程の解析に関する研究を行っています。リガンド結合のパスウェイを求めるため、リガンド結合に関わるトラジェクトリからリガンドの動きを解析しました。いくつか典型的なパターンについて計算していますが、次の図は、レバンスクラーゼにスクロースが結合する様子を解析した結果を示しています。タンパク質表面に沿ってリガンドが結合サイトに移動しているのがわかります。
フォールディングシミュレーション
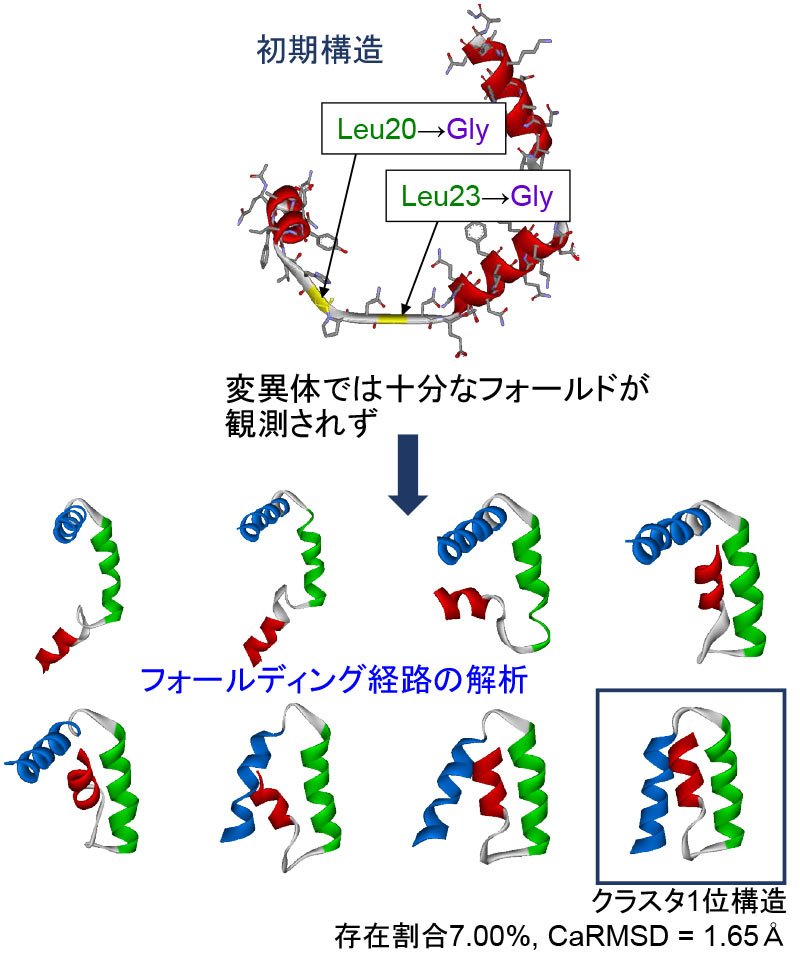
タンパク質のフォールド過程を再現するには膨大な計算時間を必要としますが、私たちは、効率的な構造サンプリングや溶媒効果の計算法、2次構造を拘束するなどの手法を用いて、シニョリン、プロテインAのBドメイン、WW ドメイン、FSD-EYなど、さまざまなタンパク質のフォールディングに成功しています。シニョリンについては、変異実験、NMR測定なども行って、フォールディングの経路を詳細に解析しました。
タンパク質の機能の解析
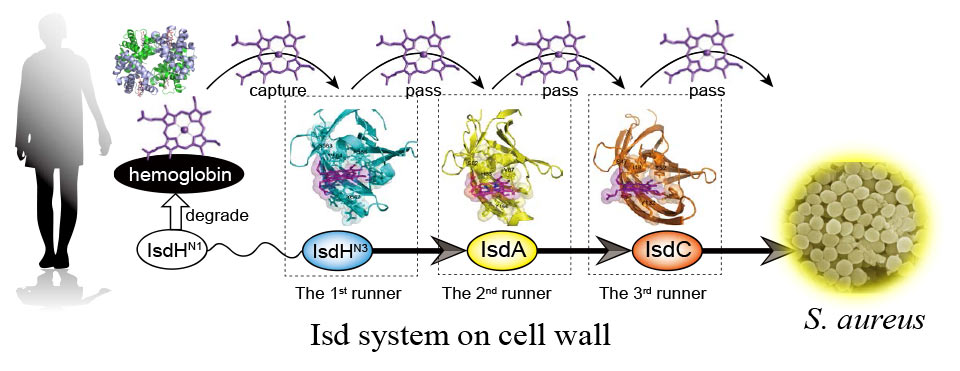
黄色ブドウ球菌のヘム輸送機構の解明
IsdH-N3, IsdA, IsdCは黄色ブドウ球菌封じ込めのターゲットタンパク質として研究されています。これらのタンパク質の構造にそれほど違いはありませんが、ヘムはIsdH-N3 → IsdA→ IsdCの順に輸送され、ヘムに対し異なる親和性を示します。私たちは、計算と実験の連携により、親和性に違いが生じる原因をタンパク質の構造をもとに調べる研究を行っています。本研究は、東京大学の津本浩平先生との共同研究です。
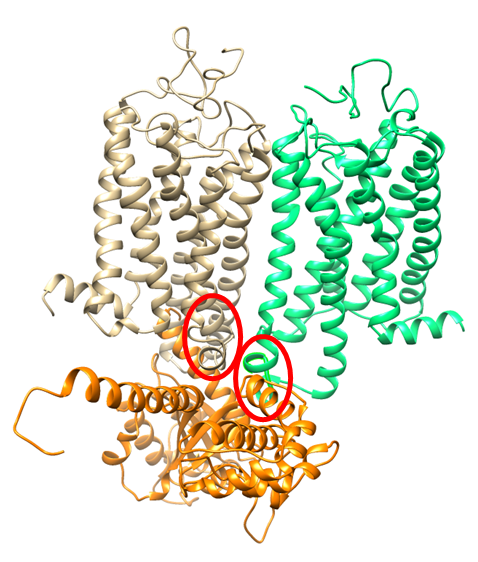
ウシ二量体グレリン受容体とGα(q)タンパク質との相互作用部位の推定
グレリンは、グレリン受容体(GHSR)に結合し、 Gタンパク質を介してシグナル伝達を引き起こします。GHSRは二量体を形成しますが、それらの細胞内loop 3のヘテロ変異個体に、とくに増体形質が見られることが知られています。本研究は、そうした変異が与える影響をタンパク質構造をもとに解析しました。本研究は、農業・食品産業技術研究機構・畜産草地研究所の小松正憲先生との共同研究によるものです。
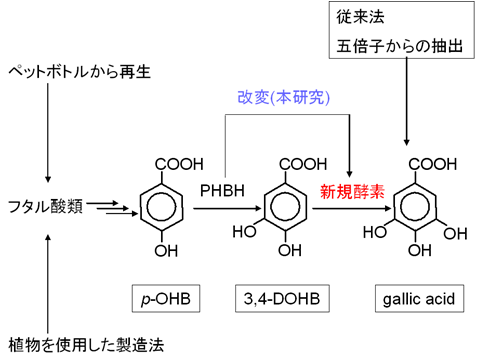
酵素改変による没食子酸の効率的な生産法の開発
没食子酸は、そのアルカリ性水溶液が還元剤として使用され、エステルの形で酸化防止剤としても使用されている工業的に有用な化合物です。本研究では、高い効率で3,4-Dihydroxybenzoate(DOHB)を没食子酸に変換するp-hydoroxybenzoate hydroxylase(PHBH)の変異体の候補を、変異体の構造モデリングと相互作用解析を行って、提案しました。本研究は、株式会社ジナリスとの共同研究によるものです。
分子動力学シミュレーションの高速化
私たちは、メニーコアシステムを用いた分子動力学シミュレーションの並列計算の研究にも取り組んでいます。