バイオインフォマティクス実習資料(平成20年度、清水謙多郎)
![]()
1. KEGGの利用
KEGGのサイト(http://www.genome.jp/kegg/)にアクセスして下さい。ゲノムネットからも簡単にたどることができます。

KEGGは総合的なデータベースですが、ここでは、「PATHWAY」データベースを使って、パスウェイの簡単な解析を実際にしてみます。上のページ中程の「PATHWAY」を指定しても、「KEGG2」(KEGG
Table of Contents)から入っても、アクセスすることができます。
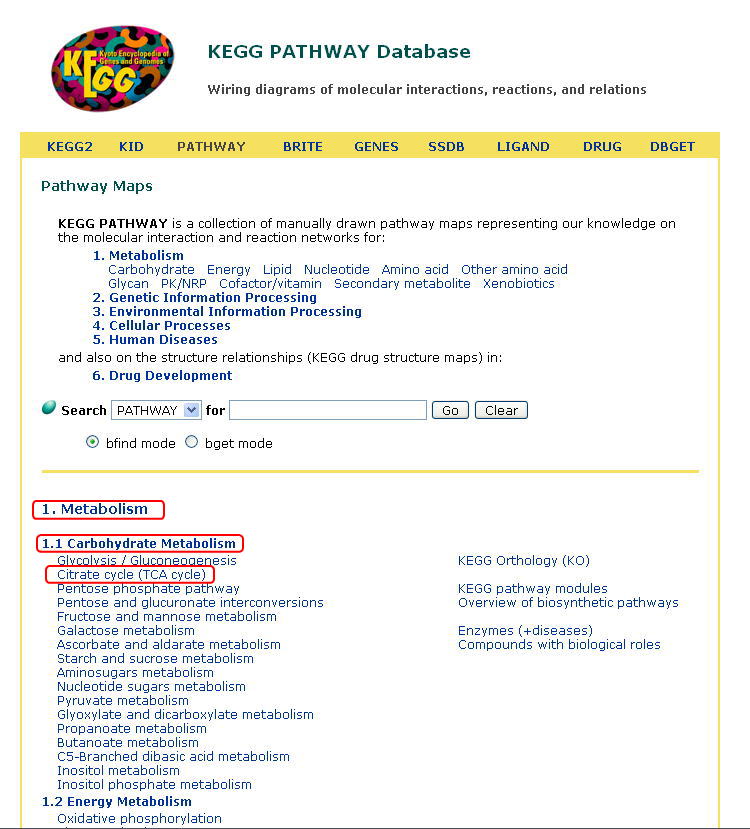
ここで、KEGGの代謝パスウェイの階層を見ながら、クエン酸回路(TCAサイクル)を表示します。「Metabolism」(代謝系)、「Carbohydrate
Metabolism」(糖代謝)、Citrate cycle(クエン酸回路)を順に見ていきましょう。
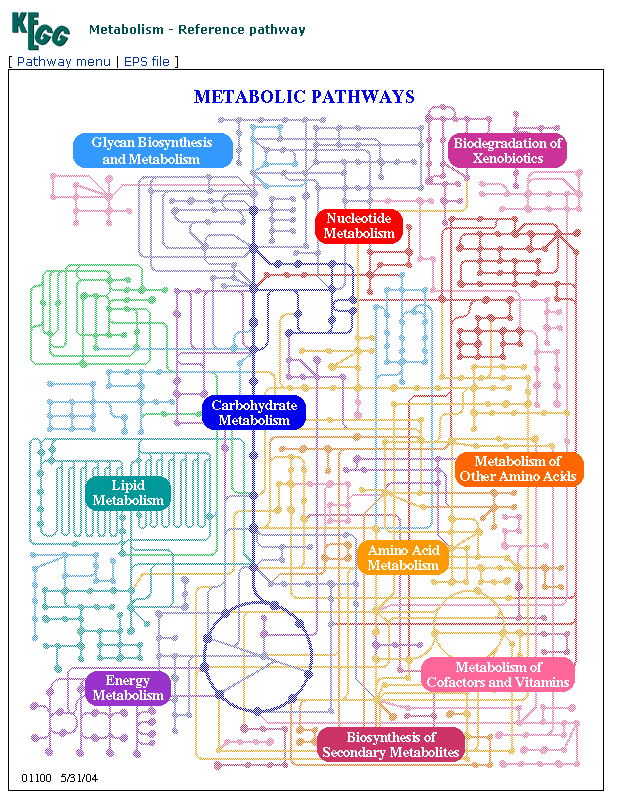
代謝系の図で、「Carbohydrate Metabolism」を選択してもかまいません。
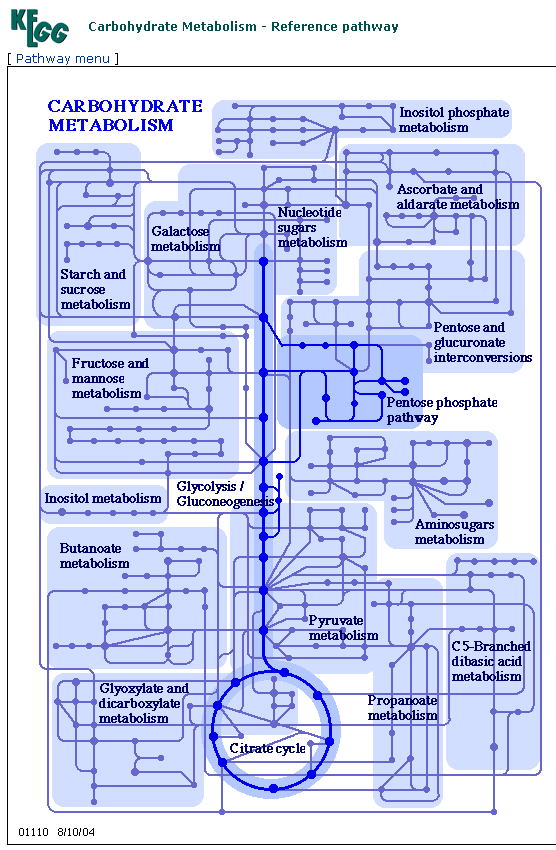
ここで、「Citrate cycle」を選択してもすることもできます。クエン酸回路のリファレンスパスウェイは以下の通りです。
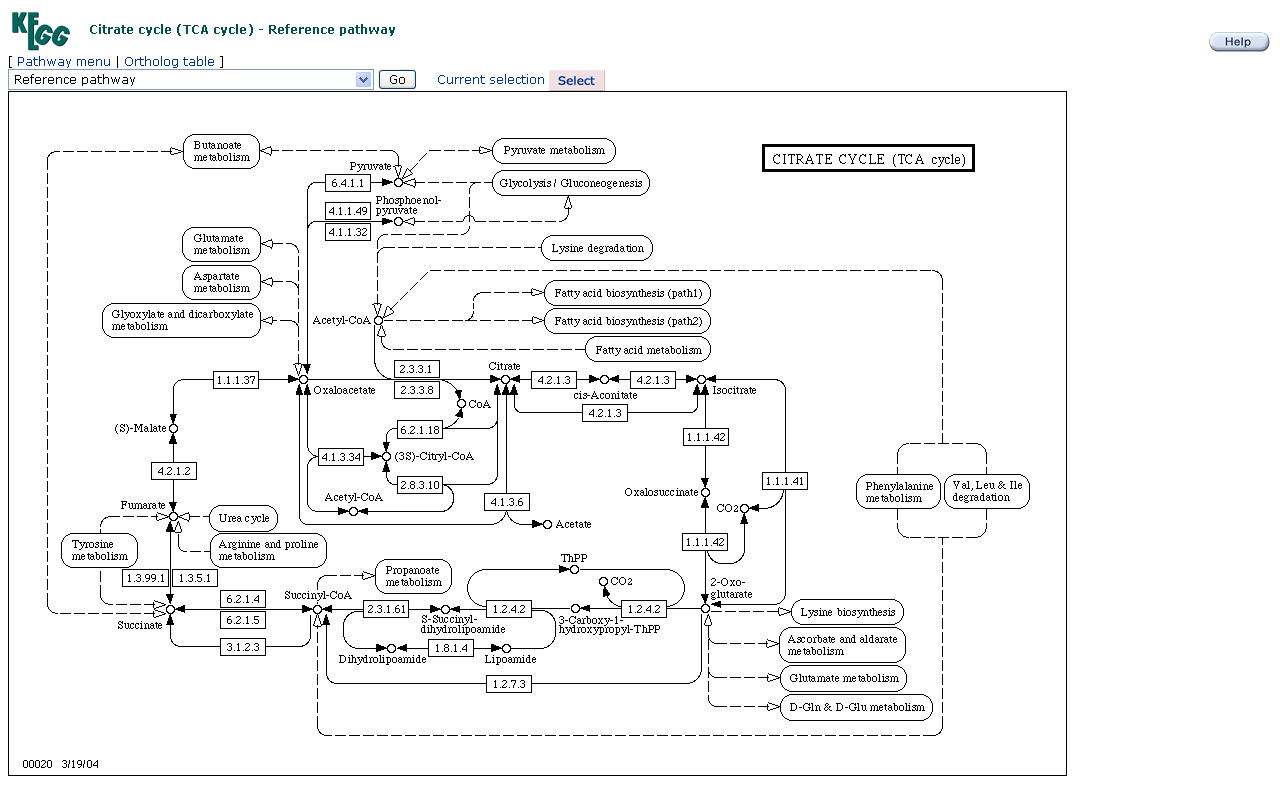
このレベルにくると、酵素や化合物など、具体的な要素が登場してきます。□はタンパク質やRNA、○は化合物を表しています。リファレンスパスウェイは、多くの生物種のパスウェイをまとめたもので、M.
Gerhardの編集による"Biochemical Pathways: An Atlas of Biochemistry and Molecular
Biology", John Wiley & Sons. によるものです。ここで、生物種ごとのパスウェイを見てみましょう。左上で、ヒト「Homo
sapiens (human)」を指定します。ヒトがもつ要素がグリーンで表示されます。できれば、他のいくつかの生物種で試してみましょう。
KEGGでは、多くの生物種で保存されている遺伝子をオーソログテーブルという表に登録しています。オーソログテーブルは、左上の「Ortholog
table」から参照することができます。生物種として「All organisms in KEGG」を指定する(選択した後、「Go」ボタンを押す)と、KEGGでアノテーションがなされている(全ゲノムが解読されている)生物種がもつ要素が表示されます。赤色がオーソログテーブル
が作成されているもの、青色が、KEGGでアノテーションがなされている生物種のうち少なくとも1つがもっているものを表しています。また、それぞれの要素をクリックすると、その詳細を知ることもできます。
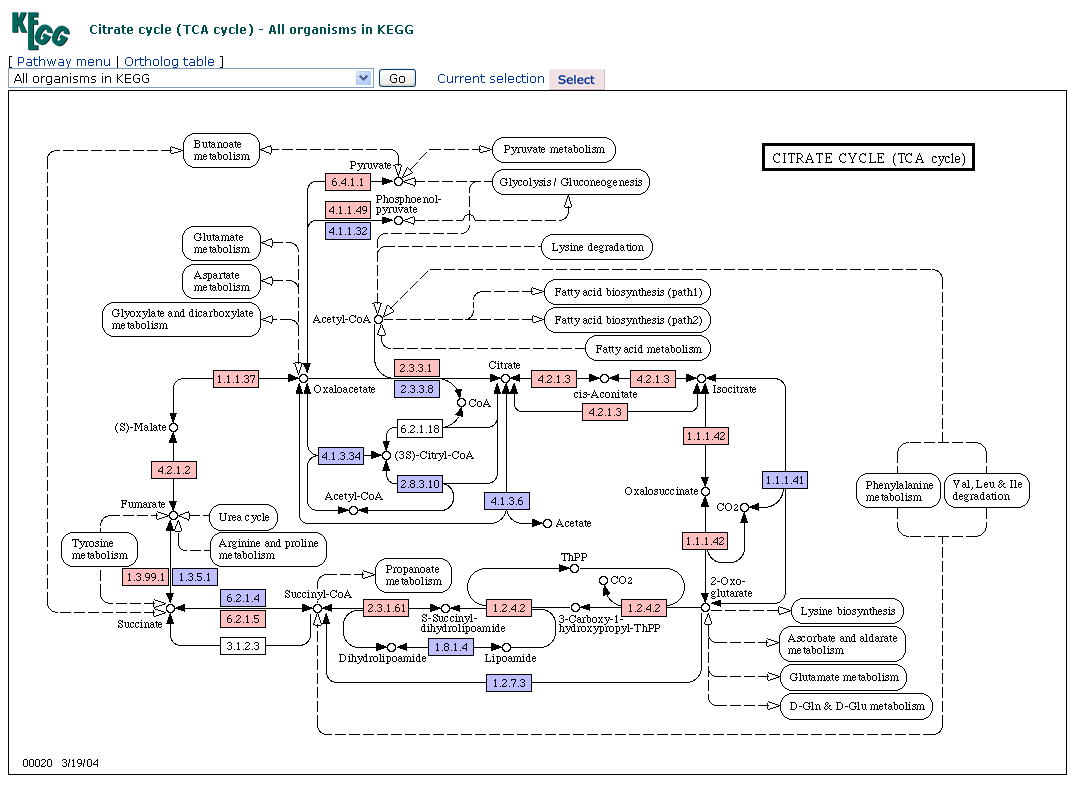
次に、生物種として緑膿菌(Pseudomonas aeruginosa PA01)を指定すると、緑膿菌ゲノムから予測された酵素は緑色で表示される。
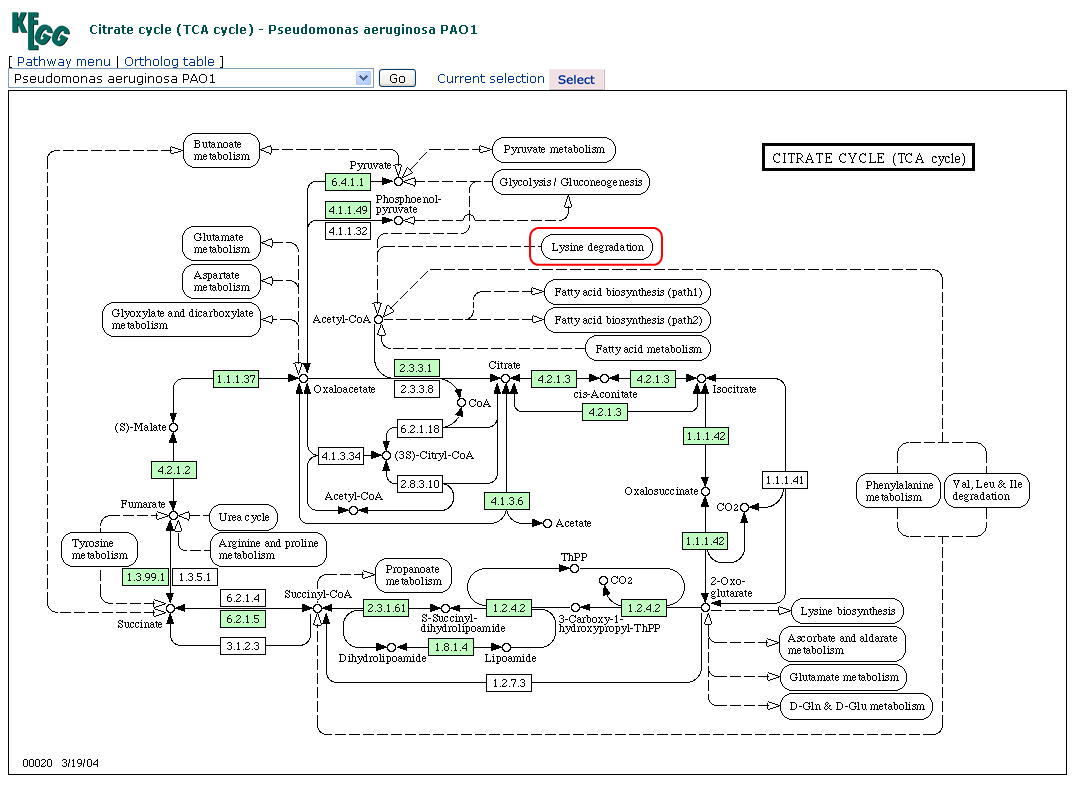
ここで、パスウェイの図の上部にある「Lysine degradation」(リジン分解系)をクリックして下さい。以下のようなパスウェイが表示されます。
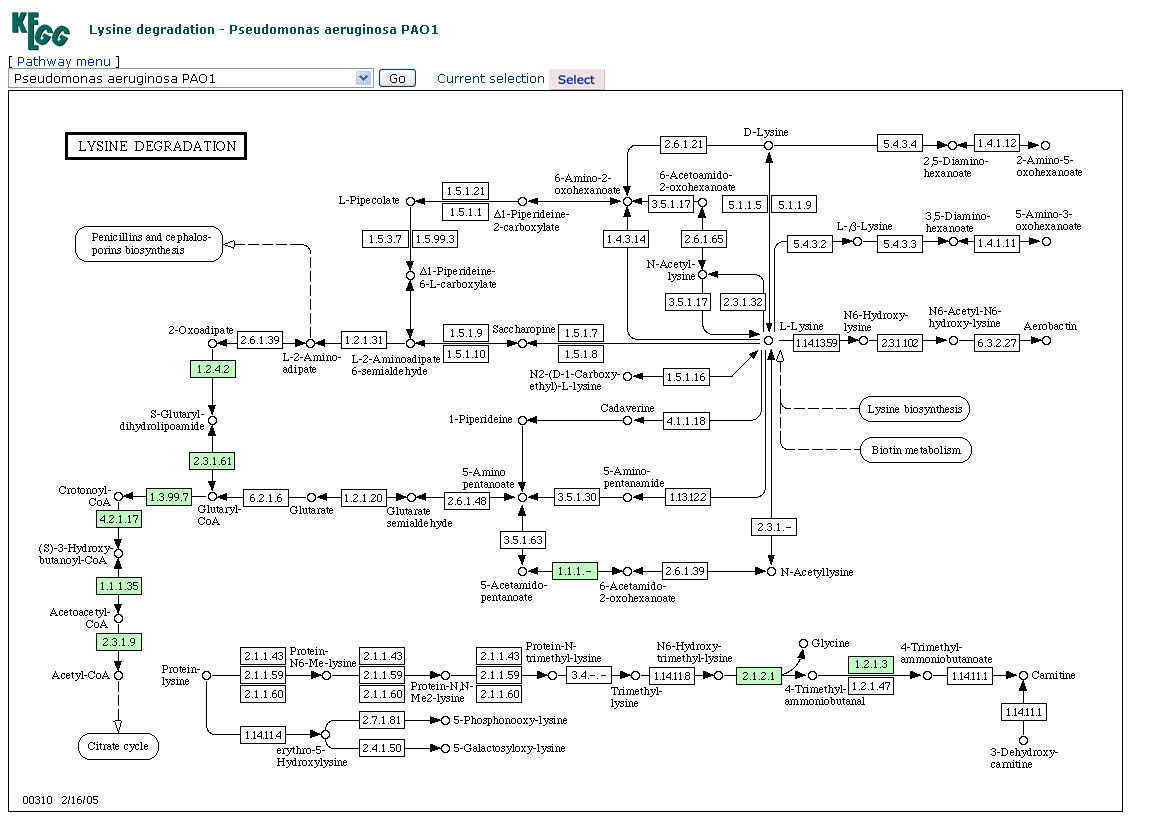
リジン分解では、リジンがいくつかのステップでアセチルCoAまで分解されてクエン酸回路に利用されるが、リジンからグルタリルCoAまでの経路の途中にある酵素が緑色になっていないことに注意して下さい。生化学実験では、緑膿菌はリジン分解系をもつことが知られているので、経路の途中で働く遺伝子の機能がまだ明らかになっていないことを示しています。
ある生物種のゲノムが決定されたとき、どのような代謝系をもつかを、既知の生物種の代謝系との比較で調べることが可能です。例えば、遺伝子の名前、酵素番号を指定し、検索することができます。生物種間のパスウェイの比較を試みてみましょう。まず、準備として、xgenes.txtというファイルを講義のページからダウンロードして下さい。このファイルは、微生物ゲノムの比較解析のためのデータベースであるMBGD(Microbial
Genome Database for Comparative Analysis)(http://mbgd.genome.ad.jp/)で、オーソログ関係にある系統プロファイルをもとに、デフォルトの微生物の集合に対してクラスタリングを適用した結果、Cyanobacteria(藍藻類)に共通に見られた遺伝子群です。これは、MBGDのトップページの「Create/View
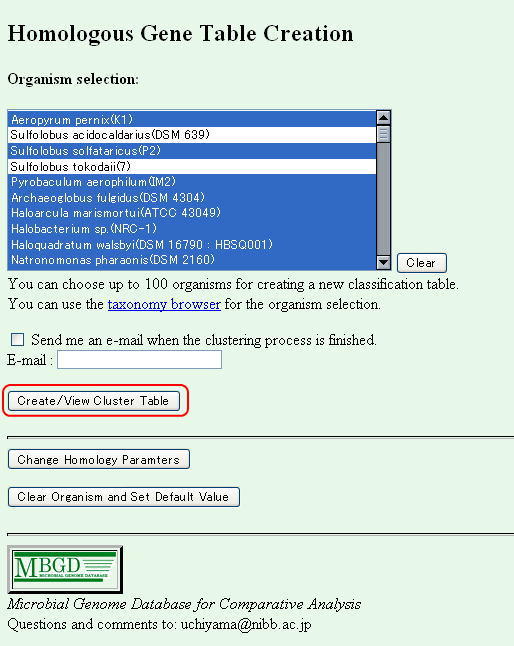
Ortholog Table」をクリックし、デフォルトの微生物をそのまま適用して得られます。
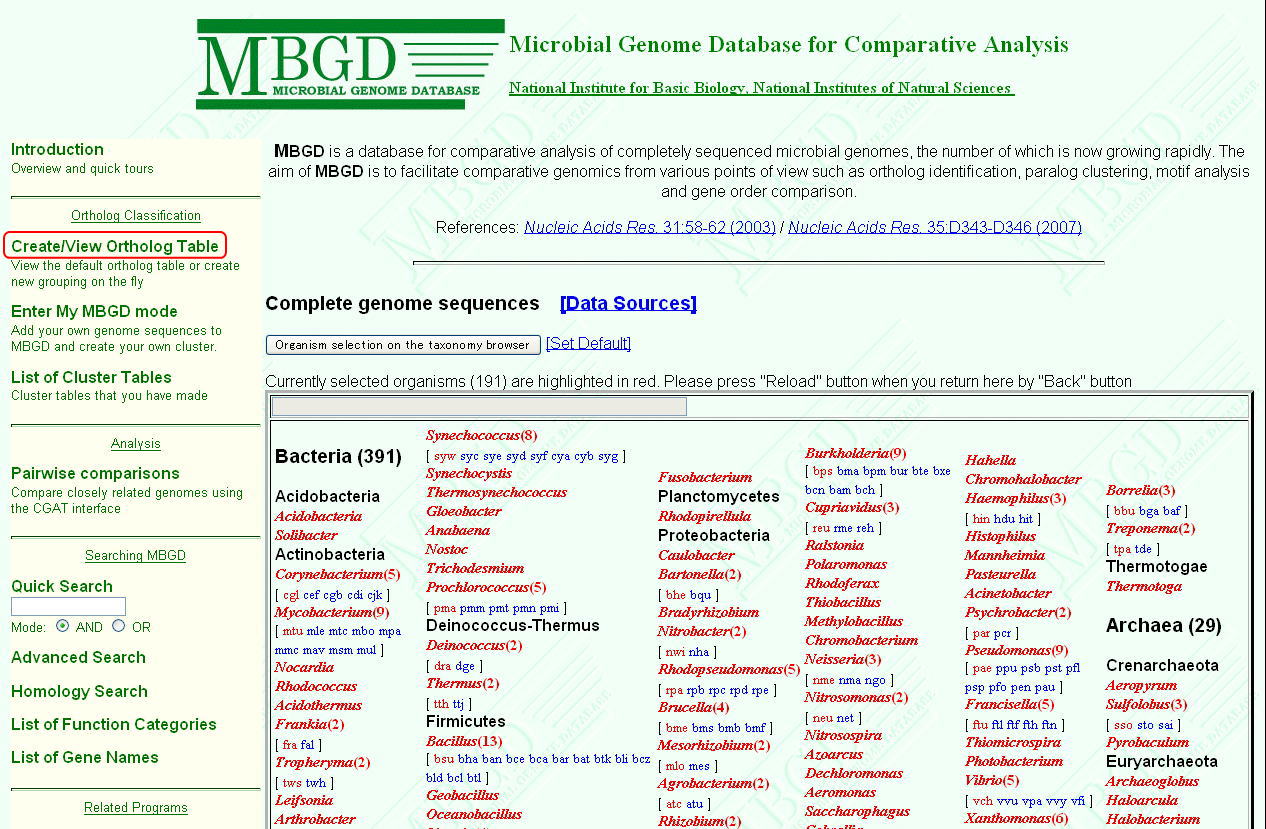
次の画面で「Create/View Cluster Table」を指定します。
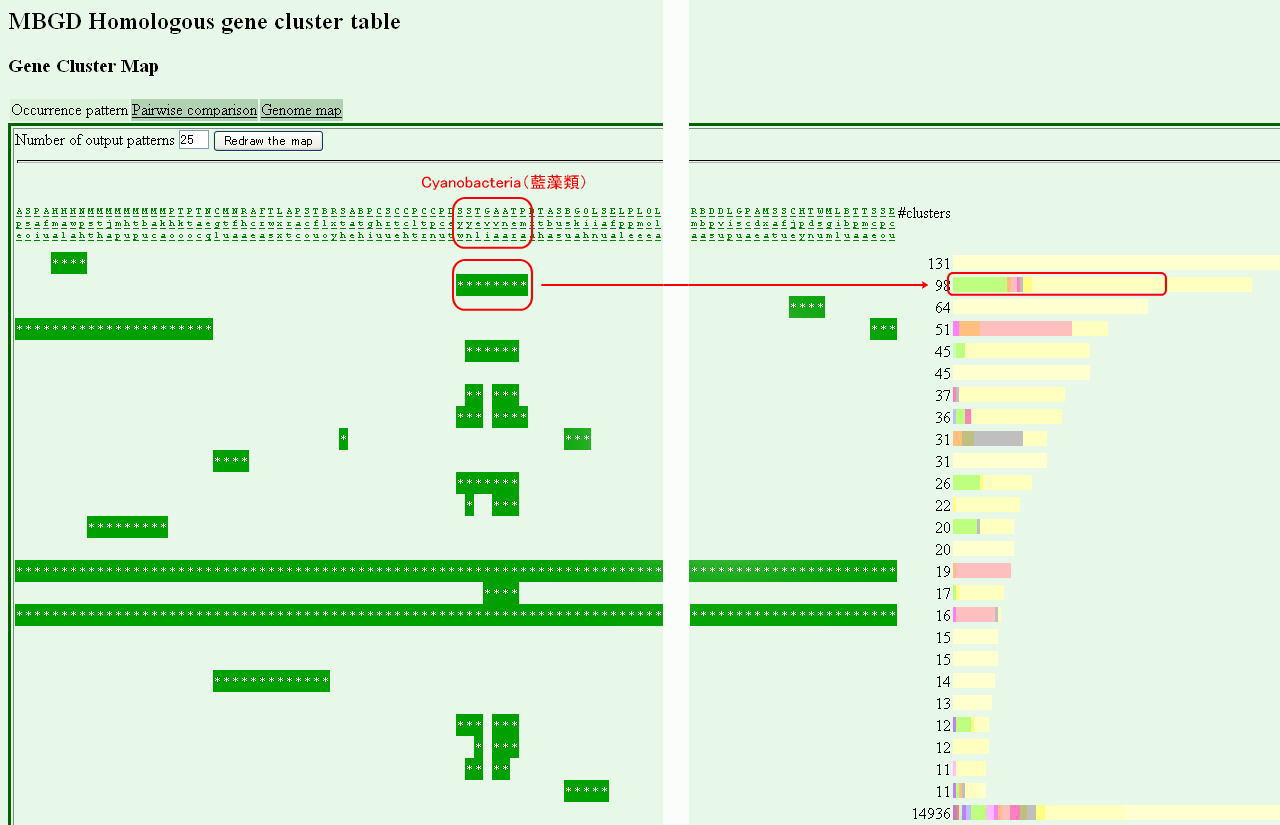
次のようなクラスタテーブルが得られます。
ここで、KEGGのトップページから「KEGG2」(KEGG Table of Contents)のページにアクセスして下さい。
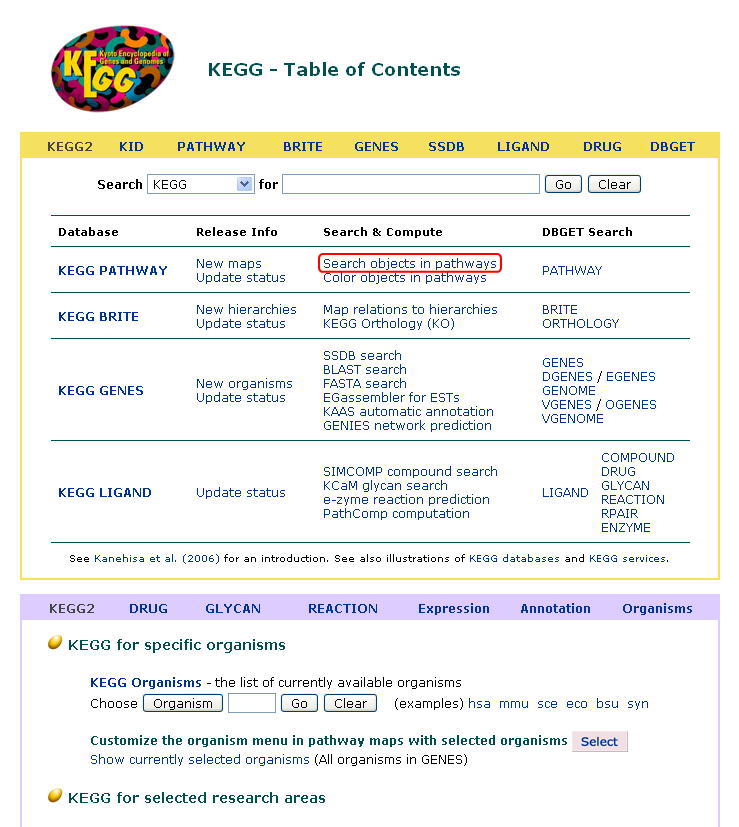
「PATHWAY」データベースに対して「Search objects in pathways」を指定します。
検索対象(「Search against」)としては、近縁の生物種の方がより比較が行いやすいと考えられますので、ここでは、藍藻類の一つであるSynechocystis
sp. PCC6803を指定します。
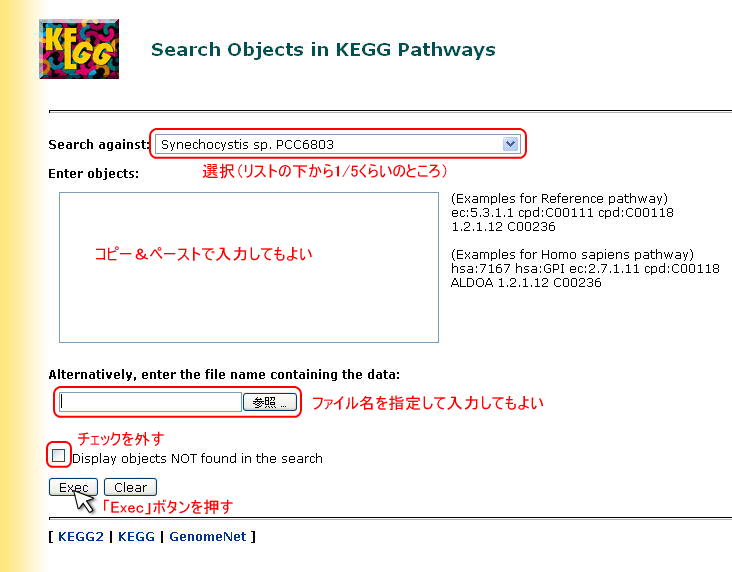
その結果は、次のようになります。

これを見てわかるように、光合成(photosynthesis)に関わる遺伝子が多いことがわかります。「KEGG2」(KEGG Table of Contents)のページで、「Color obkects in pathways」を選択しても同様の結果が得られますが、検索の結果得られた代謝系の中で、検索にかかった遺伝子を指定した色で表示することができます。