バイオインフォマティクス実習資料(平成20年度、清水謙多郎)
![]()
1. GenBankの利用
NCBIのサイト(http://www.ncbi.nlm.nih.gov/)にアクセスして下さい。以下のような画面が表示されます。
ここで、上のメニュー欄の「BLAST」を選択して下さい。
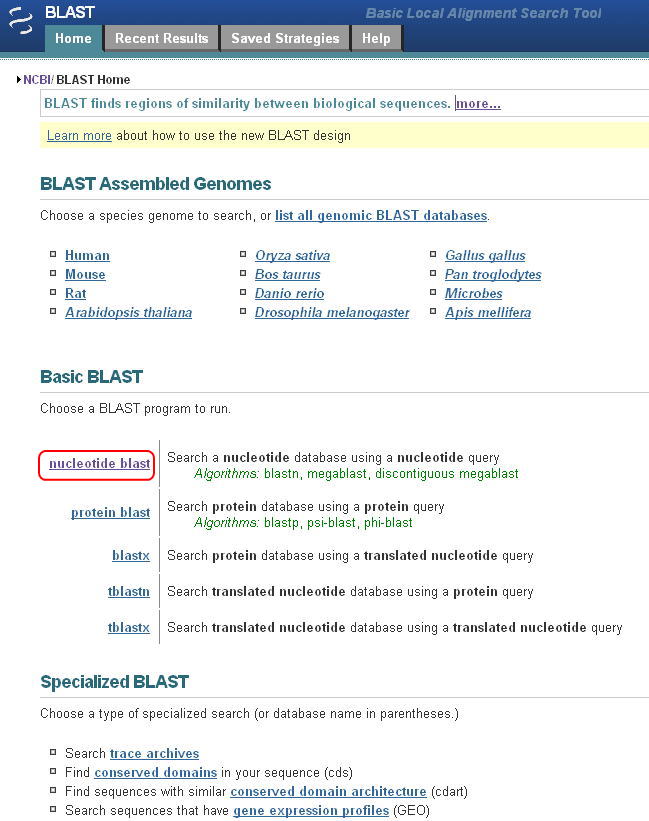
以下のような画面が表示されます。そこで、「nucleotide blast」を選択します。
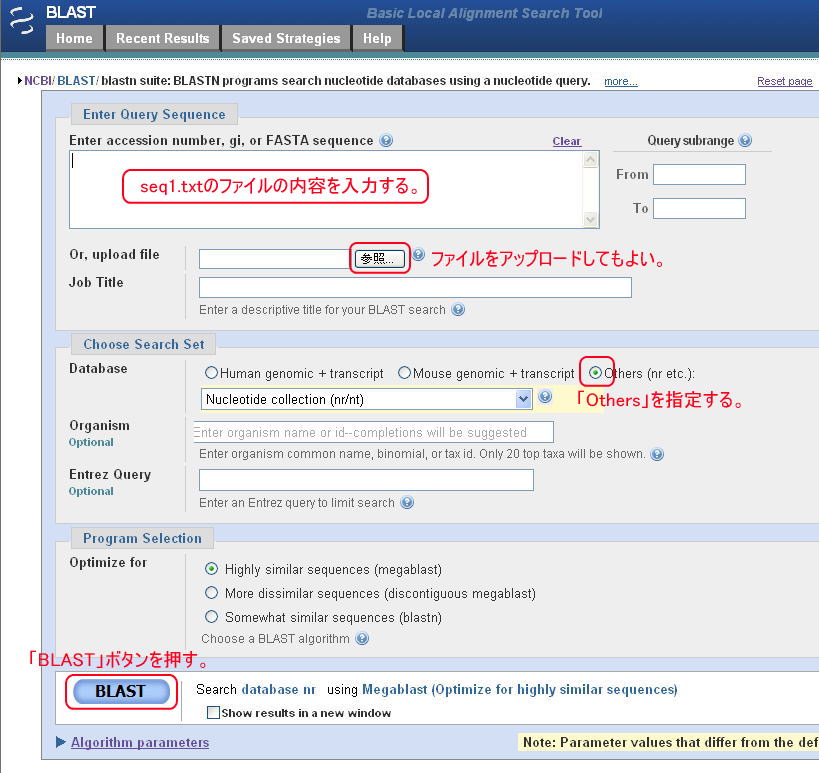
講義のページから、講義資料で使用したDNA配列の一部(seq1.txt) をダウンロードし、適当な名前を付け(seq1.txtのままでかまいません)使用しているPCに格納して下さい。メモ帳などでファイルを開き、内容をそのままコピー&ペーストして、「Search」の枠に入れても、ファイルを直接アップロードしても結構です。「BLAST」ボタンを押します。
以下の検索結果が表示されます。
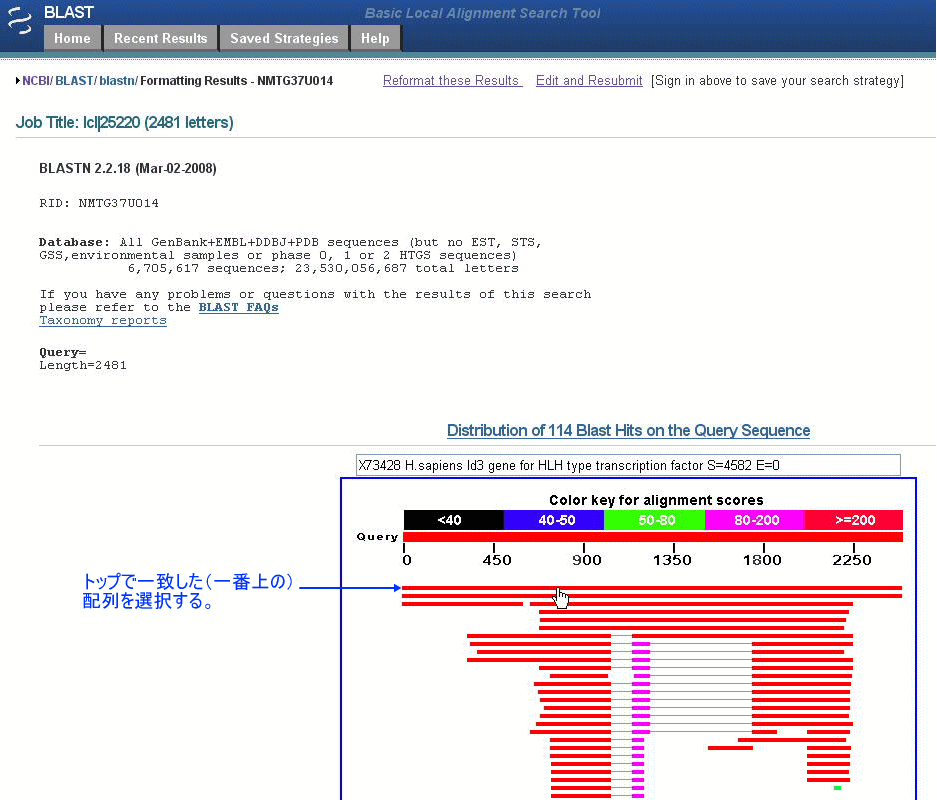
以下のようなアラインメントが表示されるので、配列のIDをクリックして、GenBankのエントリーを表示します。
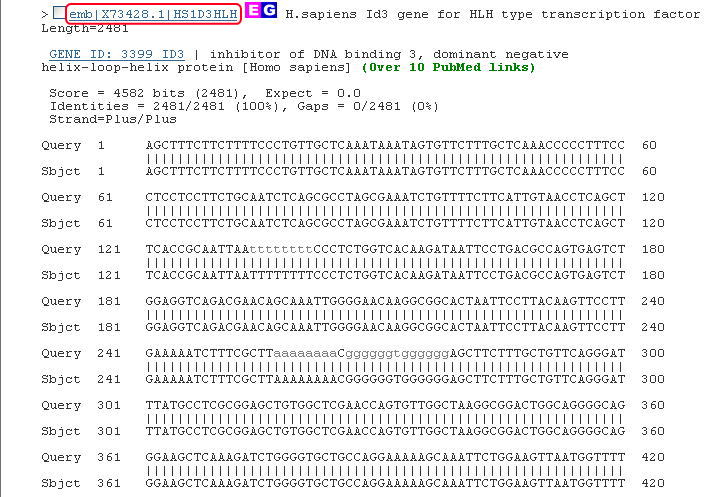
GenBankのエントリは以下の通りです。
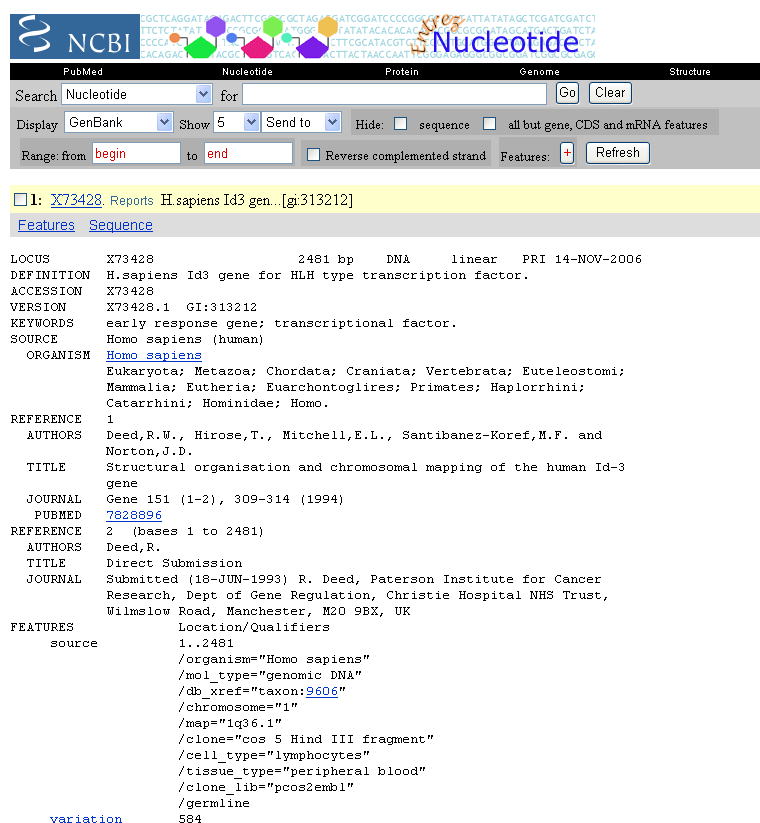
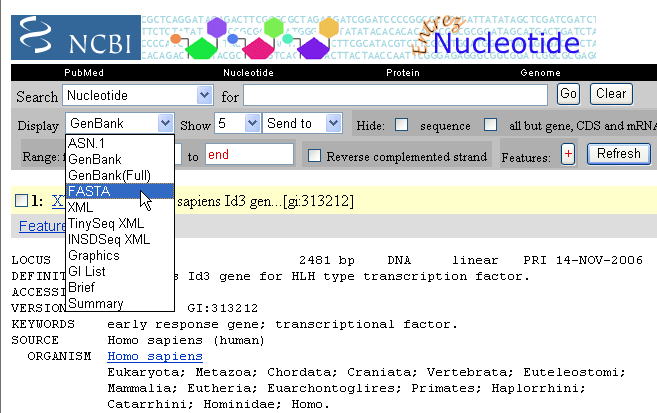
ここで、このエントリ(GenBank X73428)の塩基配列を取得します。配列を取得するには、左上の「Display」で「FASTA」を指定します。
(この配列は、次のGenScanの実行に使います。GenScanの入力では、数字は無視されるため、このエントリの下に表示されている塩基番号付きの配列をそのままコピー&ペーストして取得してもかまいません。)
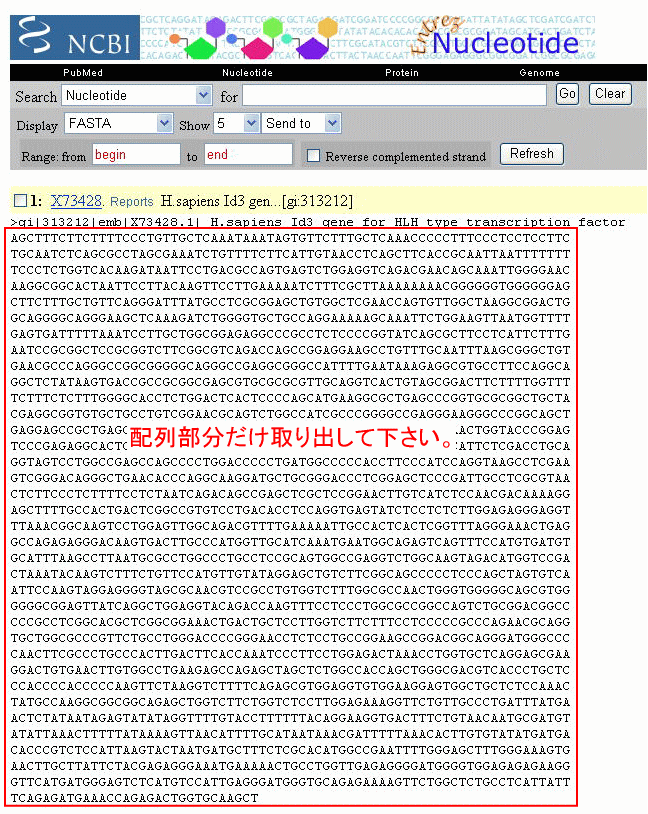
FASTA形式の配列部分だけを取り出して下さい。
2. GenScanの実行
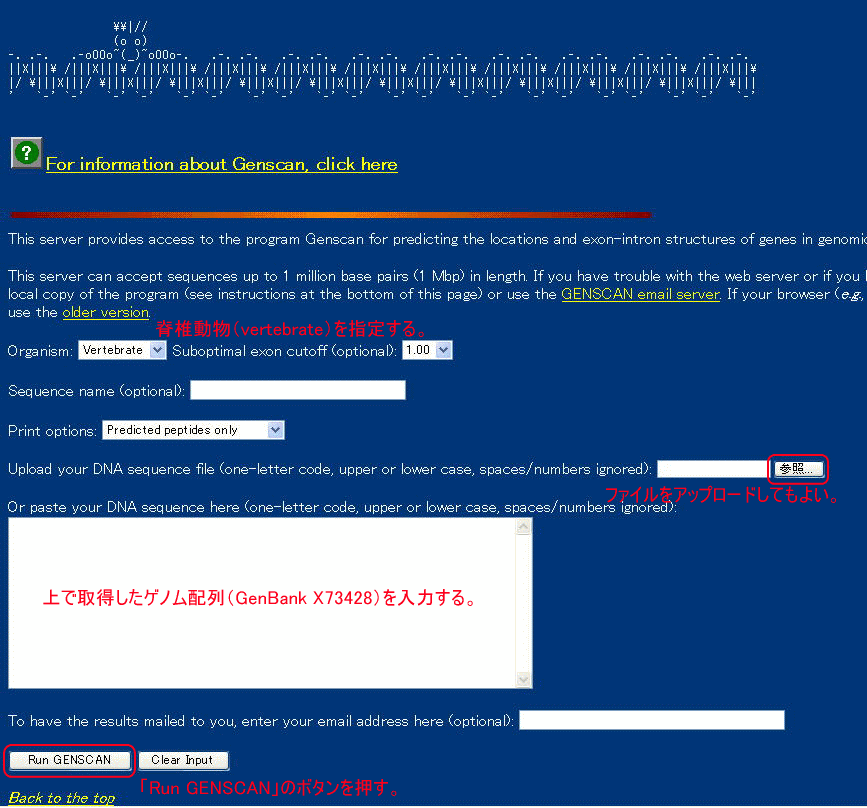
GenScanのサイト(http://genes.mit.edu/GENSCAN.html)にアクセスして下さい。以下のような画面が表示されます。

ここで、先ほどのDNA配列の一部(seq1.txt)を入力します。
以下のような予測結果が表示される。
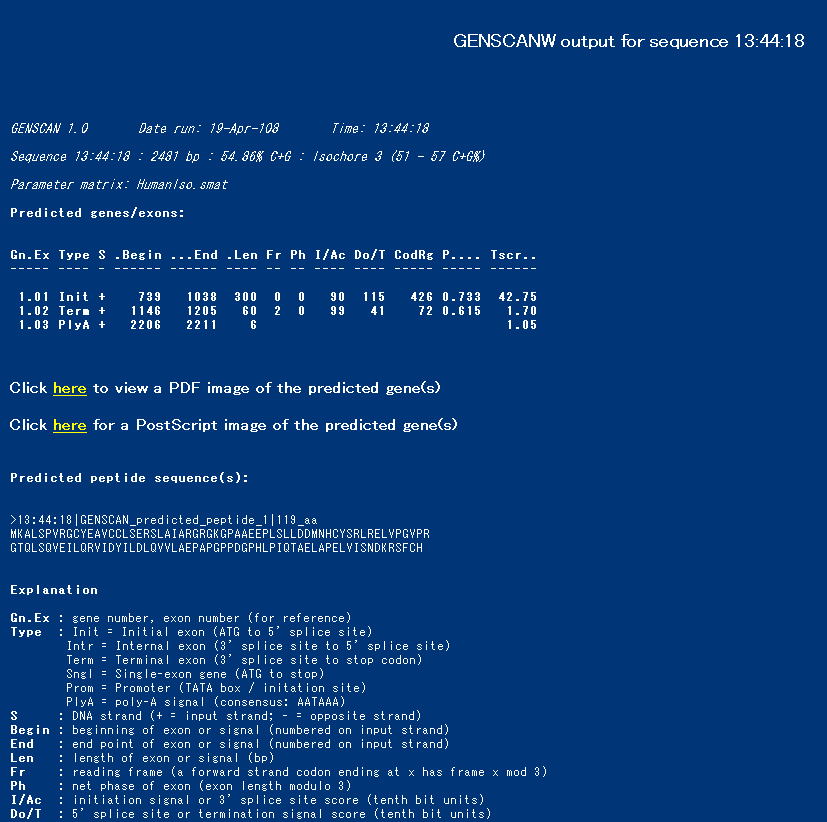
この結果と、GenBankのアノテーション(Features)の内容を比較してみましょう。
同様に、DNA配列の一部(seq2.txt)についても、GenScanで遺伝子の位置を予測して下さい。この配列は、シロイヌナズナ(Arabidopsis thaliana)のものなので、生物種を「Arabidopsis」に指定します。

この配列(seq2.txt)をBLASTで検索すると、以下のような結果が得られます。
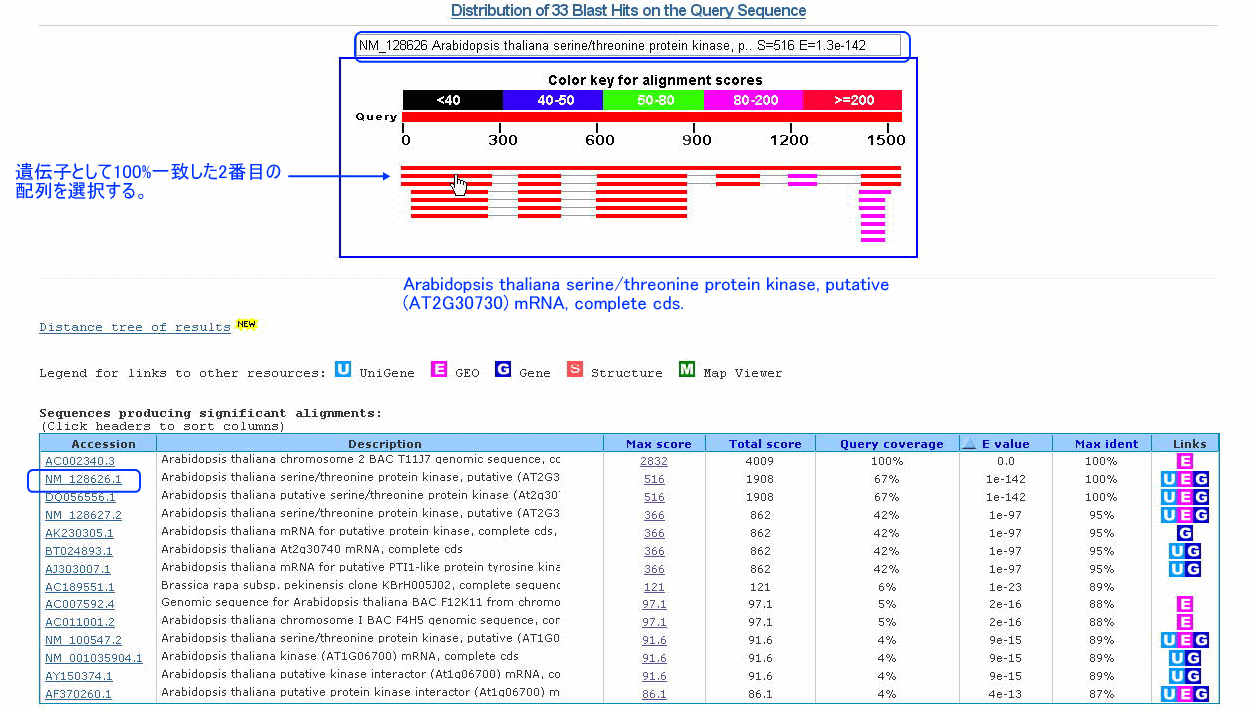
ここで、TIGRデータベースのシロイヌナズナゲノムプロジェクトのページ(http://www.tigr.org/tdb/e2k1/ath1/)を見て下さい。「Sequence Search」で配列(seq2.txt)を入力したり、「Locus Search」でAT2G30730を入力するなどして、検索を試みて下さい。

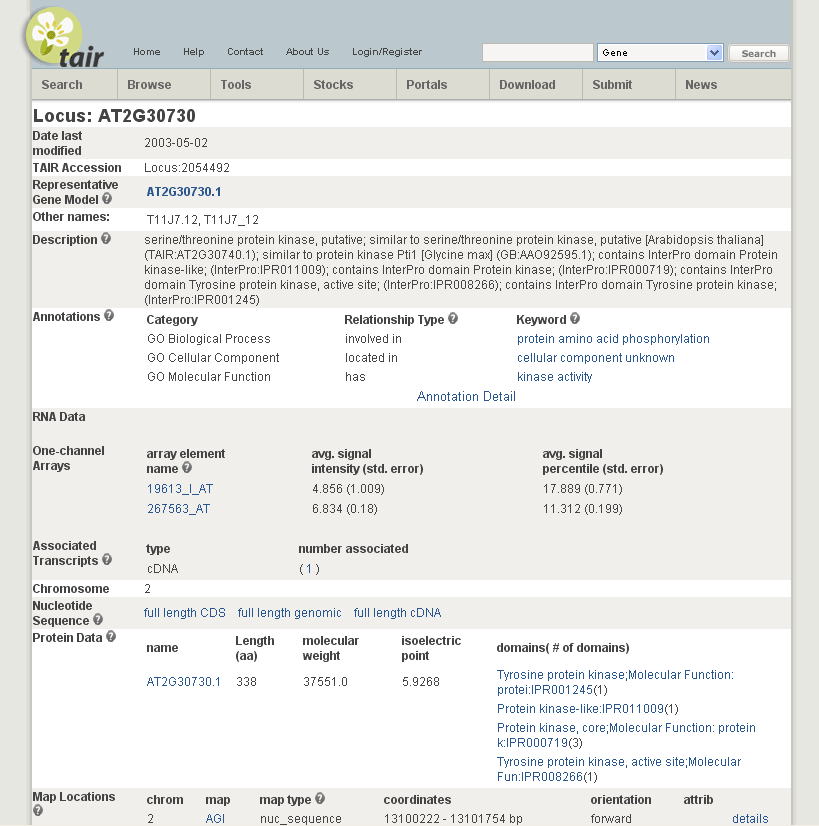
ローカスAT2G30730のエントリーは以下のようになります。ここで、「TAIR」の情報を表示してみて下さい。
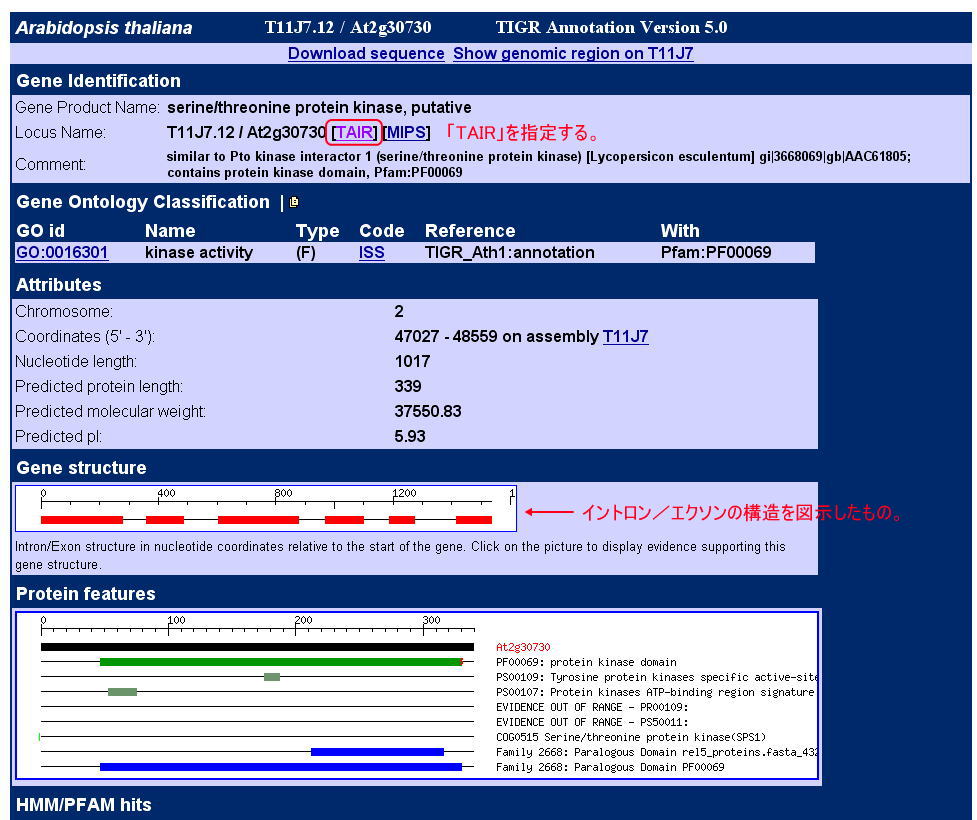
「TAIR」のページの中に、先ほどのイントロン/エクソン構造の具体的な情報が表示されているので、上で求めたGenScanの予測結果と比較してみて下さい。
GenScanによる予測で、生物種を「Arabidopsis」ではなく、「Vertebrate」にすると結果がどうなるか、試してみましょう。