タンパク質の相互作用予測
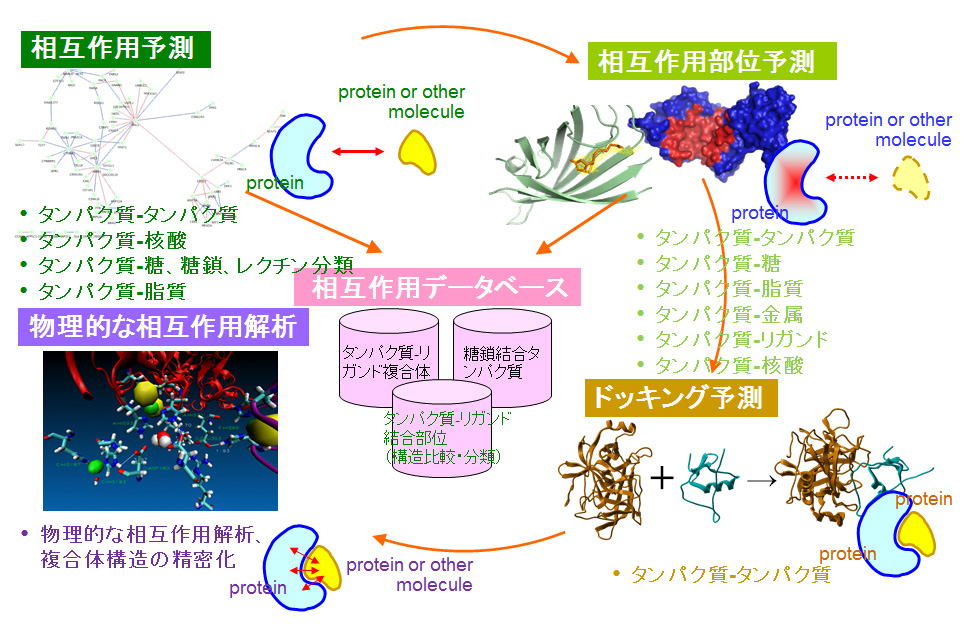
私たちの研究室では、タンパク質の相互作用について、
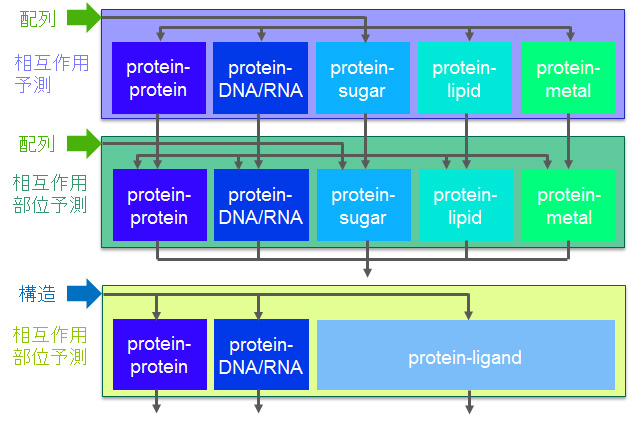
- 相互作用予測: 与えられたタンパク質が他の分子と相互作用するかどうかを予測
- 相互作用部位予測: 与えられたタンパク質の中で他分子と結合する残基はどれか、タンパク質の構造が得られている場合は結合部位がどこかを予測
- ドッキング予測: 複合体の構造を予測
の3つのアプローチから研究を行っています。また、分子シミュレーションなどを用いて、物理的な相互作用を解析する研究も行っています。
- 総合的なタンパク質-リガンド結合部位予測システム
- 機械学習を用いたタンパク質の相互作用部位予測
- タンパク質-リガンド結合部位予測ツールの自動生成
- タンパク質の構造に基づくリガンド結合部位予測
- タンパク質-タンパク質のドッキング予測
総合的なタンパク質-リガンド結合部位予測システム
私たちの研究室で開発した、あるいは開発中のタンパク質相互作用予測/相互作用部位手法を統合し、与えられたタンパク質に、タンパク質、核酸、糖、脂質、金属が相互作用するかどうか、相互作用する場合にはどこが相互作用部位となるかを一度に予測できるようにしたシステムを構築しています。
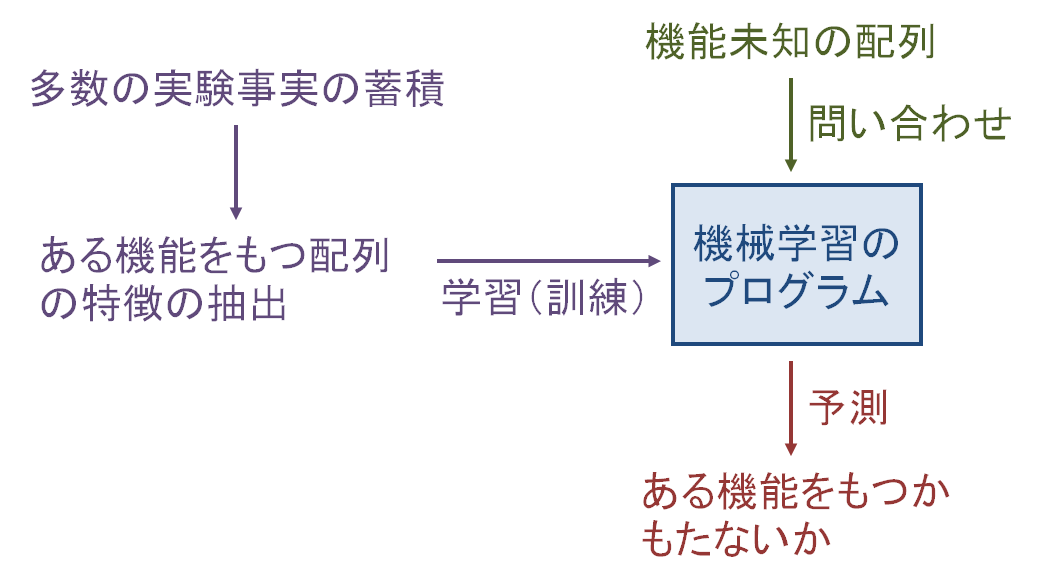
機械学習を用いたタンパク質の相互作用部位予測
タンパク質の配列から相互作用するかどうか、ある機能をもつかどうかを機械学習を用いて予測する手法を開発しています。
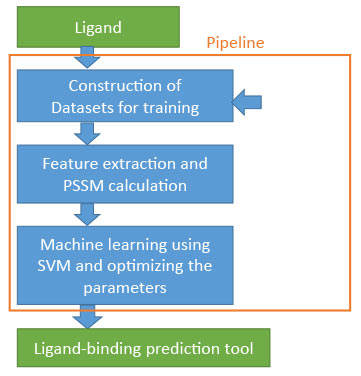
タンパク質-リガンド結合部位予測システムの自動生成
指定したリガンドの結合部位を予測するツールを自動生成するシステムを開発しています。PDBに登録された構造から、そのリガンドの結合部位を自動収集し、それらのマルチプルアラインメントを求め、PSSMを計算します。このPSSMを機械学習プログラムSVMの入力として与えて、リガンド結合部位を予測します。
現在、半日~1日で簡単に予測ツールが生成できます。また、本システムは、さらなる解析、改良の基盤としても利用できます。作成したツールは、統一的なインタフェースで利用でき、ツールの再利用を可能にします。
タンパク質の構造に基づくリガンド結合部位予測
タンパク質に低分子リガンドが結合する場合に、そのタンパク質のどの部位に低分子リガンドが結合するのかを予測する手法を開発しています。タンパク質周囲に多数のプローブ点を置き、タンパク質との相互作用エネルギーが小さいプローブ点のクラスタを求め、さらに配列保存度を考慮して予測します。
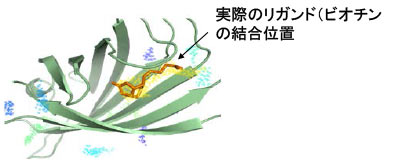
次の図は、ストレプタビディンにリガンドのビオチンが結合する位置を予測した結果です。ドットは、タンパク質構造の周辺に格子状に配置したプローブ点のうち、タンパク質との相互作用エネルギーが小さいものをクラスタリングしてまとめたものです。青色から黄色に近づくほど予測順位が高く、図では、第1位と予測した位置(黄色)が、実際のビオチンの結合位置と一致していることを示しています。
タンパク質-タンパク質のドッキング予測
ドッキングシミュレーションについては、球面調和関数と動径基底関数を用いた高速計算手法の開発を行っています。私たちは、タンパク質分子を複数のレイヤーに区切り、レイヤーごとに動径基底関数を定義し、展開次数の調整を行えるようにしており、一層の高速化を図っています。
こうしたドッキングの高速化は、アンサンブルドッキングなどによるフレキシブルドッキングの実現、網羅的な相互作用予測や基質探索に有効と考えられます。
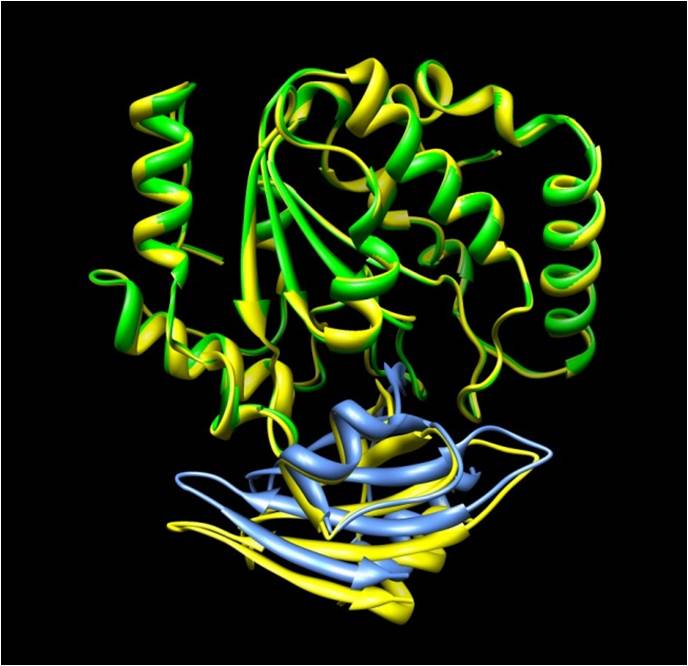
次の図は、グリコシラーゼ(緑色)とそのインヒビター(青色)とのドッキング予測の結果を表しています。黄色は、すでにわかっている複合体の結晶構造で、予測した結果と重ね合わせて表示しています。